JIPB近日在线发表了上海师范大学生命科学学院张辉团队和中科院上海植物逆境生物学研究中心朱健康团队的合作论文“Efficient generation of homozygous substitutions in rice in one generation utilizing an rABE8e base editor”(https://doi.org/10.1111/jipb.13089)。该工作开发的腺嘌呤碱基编辑器rABE8e,在水稻绝大多数靶位点的编辑效率几乎为100%,且在T0代即可高比例获得纯合碱基替换的材料。
近年来开发的腺嘌呤碱基编辑器ABE,可实现体内基因组特定序列上A/T到G/C的定点替换。为了提高ABE的效率,研究人员已经进行了多方面的改进,如优化核定位信号(bpNLS)和密码子使用、用脱氨酶单体代替二聚体、采用进化的脱氨酶变体等。但目前植物中最新一代的腺嘌呤碱基器ABEmax,在大多数位点的编辑效率仍低于50%,且很难在植物转化当代获得纯合碱基替换的材料。
2020年,David Liu团队通过体外进化ABE7.10脱氨酶产生了新的脱氨酶变体TadA8e。基于TadA8e的ABE8e系统催化DNA脱氨的速度比ABE7.10快约1100倍,ABE8e在哺乳动物细胞中展示了比ABE7.10显著提高的编辑效率。该工作还在TadA8e上引入V106W(TadA8e V106W)用以降低脱靶效应。
本研究基于水稻ABEmax系统,将其中的ecTadA:ecTadA *7.10二聚体用植物密码子优化的TadA8e V106W单体代替,分别与Cas9n(识别NGG为PAM的序列)及Cas9n-NG(识别以NG为PAM的序列)相结合,升级为新的水稻编辑器rABE8e(rice ABE8e)。通过对水稻中的11个靶序列测试发现,rABE8e在3个靶序列的编辑效率为100%,8个靶序列的编辑效率超过80%;而ABEmax只在3个靶序列的编辑效率高于70%,另外8个靶序列的编辑效率都小于50%。更为重要的是,在编辑效率超过80%的8个位点,rABE8e产生纯合碱基替换的效率都大于60%;而ABEmax在一半(5/10)靶序列没有检测到纯合碱基替换。rABE8e 在A5和A6位点(以PAM序列作为21-23位)编辑效率和纯合替换效率最高,该系统有很高的编辑特异性,且产生的碱基替换能够稳定的遗传到下一代。以上结果表明,相比于目前广泛应用的ABEmax系统, rABE8e在靶位点编辑效率和碱基纯合替换效率方面都有实质性的提高,这将大大有利于其在基因功能研究和精准分子育种中的应用。
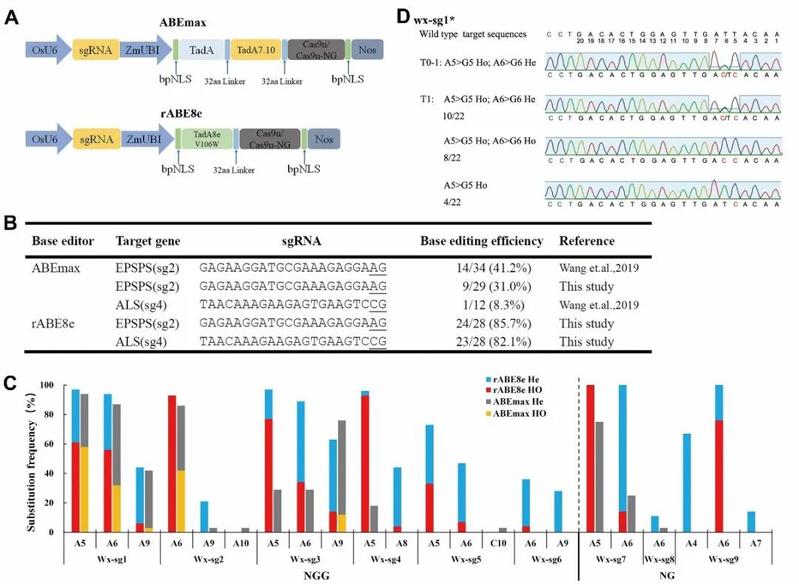
图1. rABE8e的结构和编辑特性
在本文投稿过程中,安徽农科院魏鹏程团队、中国农科院周焕斌团队和电子科技大学张勇团队等分别报道将TadA8e与不同的Cas9变体结合用于植物基因组碱基编辑,均表现出了比其它脱氨酶更高效的编辑效率。ABE8e在编辑效率上已经足以满足科研或育种的需要,后续可以在编辑框范围及特殊序列的编辑方面开展工作。
张辉团队的研究生魏闯和青年教师汪冲博士为论文的共同第一作者。张辉教授和朱健康研究员为共同通讯作者。该工作得到国家自然科学基金经费资助。张辉课题组主要利用基因编辑技术对水稻和花卉等作物进行精准性状改良。